Un polymère négativement chargé
Parmi tous les polymères hydratés de la cellule, l’ADN est sûrement celui qui retient le plus d’eau. En effet, sous forme B il contient environ 95 pds% d’eau. La vision de cette belle double hélice support de l’information génétique complètement anhydre sans sa gaine d’eau hante donc les livres de biologie. Elle donne donc l’illusion que l’eau ne joue que le rôle de bouche-trou. La réalité est tout autre, car pour lier les nucléosides entre eux, l’ADN utilise des ions phosphate négativement chargés. Or, chaque nouveau nucléotide amène deux charges négatives. La molécule d’ADN aurait donc bien peu de chances de s’autoassembler s’il n’existait pas un mécanisme de neutralisation de cette charge. Combien de biologistes savent que la durée de vie d’une double hélice dans le vide, c’est-à-dire sans eau ni compensateur de charges, est d’à peine 50 picosecondes dans le vide [1] ?
Parler d’autoassemblage sans invoquer l’eau stabilisatrice n’est pas très sérieux. Car, est la véritable génératrice de la structure en double hélice. Par conséquent, ce n’est pas une couche d’eau qu’il faut pour stabiliser la double hélice d’ADN, mais au moins 2 couches successives [2]. Ces dernières doivent aussi coopérer intimement avec des ions magnésium Mg2+. Ceci, afin d’arriver à neutraliser efficacement l’effroyable densité de charge négative qui est associée à l’énorme poids moléculaire (≈ 7 GDa) de cette molécule. En effet, la figure suivante montre l’importance cruciale du magnésium pour la neutralisation de la charge de la molécule d’ADN :
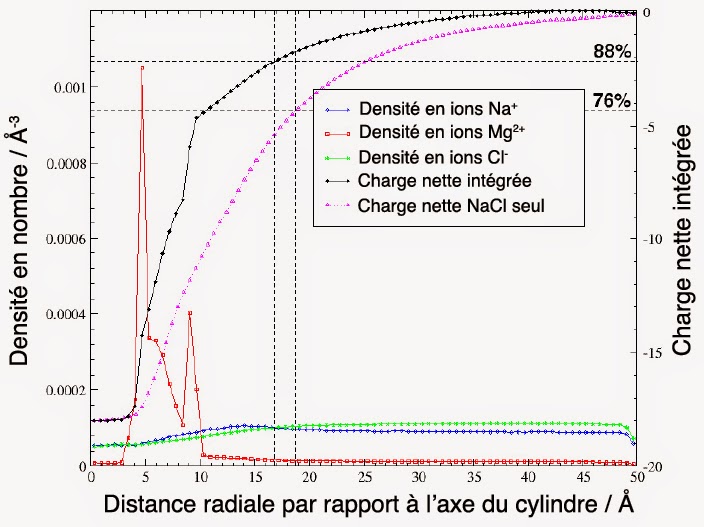
Structure de l’ADN
Il est de plus possible aujourd’hui de cristalliser non seulement les protéines, mais également l’ADN. Les méthodes de diffraction des rayons X sur ces cristaux ont été tellement perfectionnées qu’il est possible de voir ces fameuses couches d’hydratation. Ainsi, ces dernières présentent une activité de l’eau très différente de celle de l’eau liquide ou des solutions aqueuses diluées. Par exemple, pour l’ADN, on trouve que l’eau s’insinue partout où elle peut dans la double hélice. On y trouve des zones sans aucun ordre particulier. Mais, aussi des zones présentant des arrangements polygonaux. On retrouve alors, dans ces zones, une prédominance de pentagones [3] :
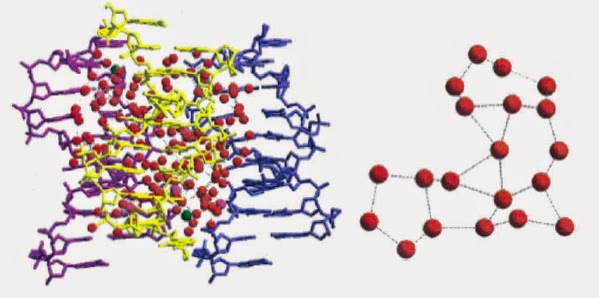
Grâce à l’excellente résolution, on a pu ainsi étudier un duplex nonamérique d(GCGAATTCG) de 433 atomes. Tout autour, on a trouvé 187 molécules d’eau dont 151 bien ordonnées. Il y avait aussi 7 ions magnésium et 1 ion chlorure. D’où un taux d’hydratation de 39 pds%. Il est toutefois important de signaler qu’in vivo ou en solution, l’ADN n’est en aucun cas dans un environnement cristallin. Donc, toutes ces structures vues par diffraction des rayons X ne préjugent en rien de la structure réelle de l’ADN intracellulaire. Des mesures d’activité de l’eau au contact de l’ADN ont ainsi montré qu’il était impossible de descendre en dessous d’une valeur aW= 0,02. Cela correspond à une enthalpie d’hydratation de l’ordre de -25 kJ·mol-1. Une telle enthalpie reste négative jusqu’à aW= 0,92 [4].
Activité de l’eau
Il est donc impossible de déshydrater complètement l’ADN. Il restera toujours au moins 4 molécules d’eau par paire de base (20 pds%). Alors qu’à l’autre extrémité l’ADN, peut se gorger d’eau sans limite [5].
Pour une activité de l’eau aW= 0,62, on trouve 10 molécules par paire de base (23 pds%). De plus, la transition ADN-A ⇄ ADN-B est observée pour aW= 0,80. Ceci correspond donc à 20 molécules d’eau par paire de base (37 pds%). Au-delà d’une valeur aW= 0,92, soit 35 molécules par paire de base (51 pds%), le temps d’équilibrage devient extrêmement long. Sur le plan dynamique, le temps moyen de résidence de la molécule d’eau autour de l’ADN est de 18 picosecondes. Ceci, pour un taux d’hydratation de 24 pds%. Il tombe ainsi à 10 picosecondes pour un taux d’hydratation de 71 pds% [6].
Ces valeurs sont d’un ordre de grandeur supérieure au temps de résidence diffusionnel mesuré dans l’eau pure qui est de 1,25 picosecondes à T = 20 °C. Au voisinage de l’ADN, on aurait donc une eau surfondue de température inférieure à -17 °C [7].
Références
[1] Gerstein M., Levitt M., «Simulating water and the molecules of life», Sci. Amer., Nov. (1998), 101-105.
[2] Ruffle S. V., Michalarias. I., Li J.-C., Ford R.C., «Inelastic neutron scattering studies of water interacting with biological membranes», J. Am. Chem. Soc., 124 (2002) 565-569.
[3] Soler-Lopez M., Malinina L., Subinara J.A ., «Solvent organisation in an oligonucleotide crystal», J. Biol. Chem.,276(2000) 23034-23044.
[4] Leal C., Wadsö L., Olofsson G., Miguel M., Wennerström H., «The hydration of DNA-amphiphile complex», J. Phys. Chem. B,108(2004) 3044-3050.
[5] Clark G. R., Squire C. J., Baker L. J., Marti R. F., White J., «Intermolecular interactions and water structure in a condensed phase B-DNA crystal», Nucl. Acids Res.,28(2000) 1259-1265.
[6] Beta I. A., Michalarias I., Ford R. C., Li J.-C., Bellissent-Funel M.-C., « Quasi-elastic neutron scattering study of hydrated DNA », Chem. Phys.,292(2003) 451-454.
[7] Teixeira J., Bellissent-Funel M.-C., Chen D. H., Dianoux A. J., « Experimental determination of the nature of diffusion motions of water molecules at low temperatures », Phys. Rev. A,31(1985) 1913-1917.


Leave a Reply