Épisode 18, Covid-19, mai 2020
Jouer avec les génomes
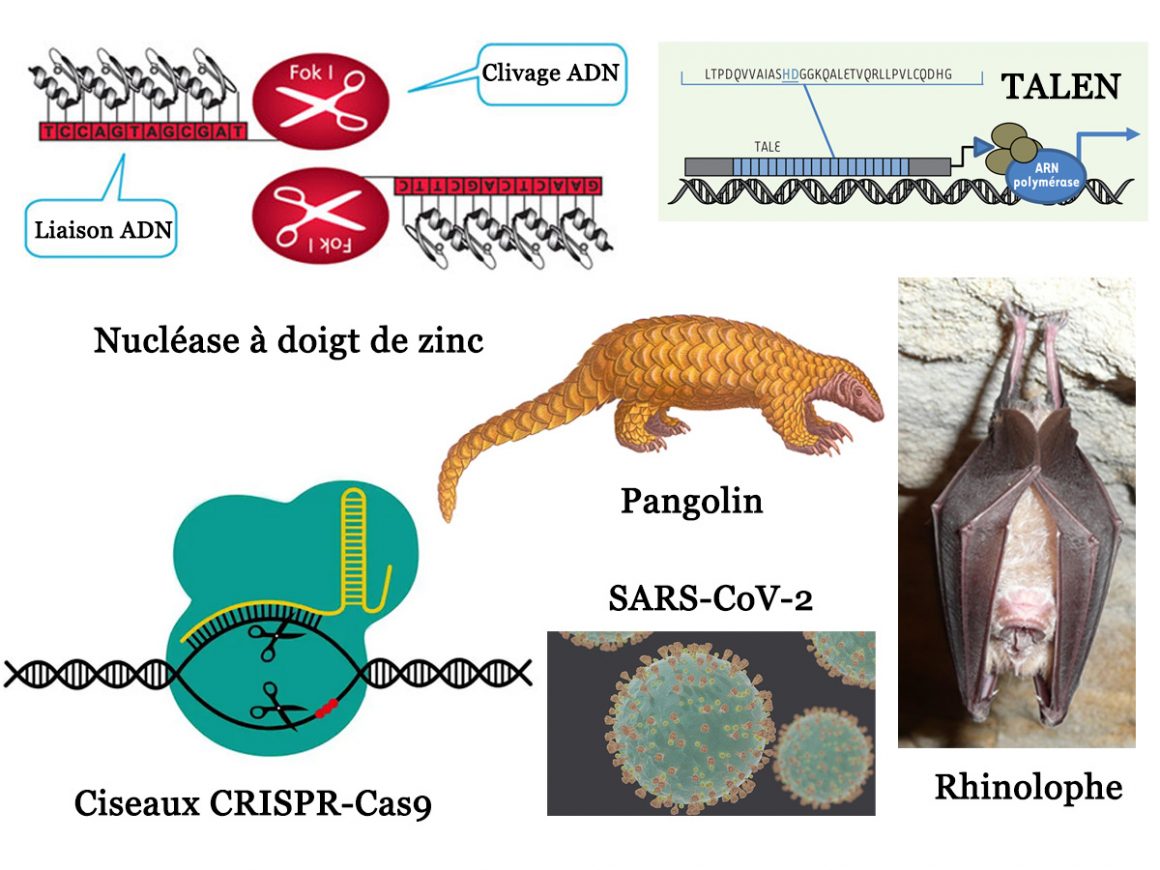
Le virus SARS-CoV-2 ne serait-il pas sorti d’un laboratoire de recherche chinois à Wuhan ? Or, l’un des enjeux majeurs des biotechnologies et de la recherche biomédicale est de faire joujou avec les génomes. Or, si les virus à ARN ont une fâcheuse tendance à muter, la science dispose de véritables ciseaux moléculaires. Ils permettent la modification précise des génomes de type ARN ou ADN (cf. chronique n°8). Les premiers ciseaux moléculaires datent de 1981, lors de la découverte d’une enzyme baptisée FokI (ou Fok-1). On les a identifiés dans une bactérie de type Flavobacterium okeanokoites (d’où l’acronyme Fok).
Cette protéine est capable de se lier, via son extrémité -amine-, à une molécule d’ADN. Elle le fait au niveau de tout site de type 5′-GGATG-3′. Cette liaison active l’autre extrémité “carboxylate” où se trouve une endonucléase de restriction. Ceci provoque le clivage spécifique de l’ADN lié. Le ciseau est aveugle. Ceci signifie qu’il coupe tout ce qui se trouve après le nucléotide le plus proche du site de reconnaissances. Il ne se préoccupe pas de ce qui se trouve dans les séquences coupées. Pour cela, il compte jusqu’à 9 sur le brin 5’->3’. Puis jusqu’à 13 sur le brin complémentaire 3’->5’. Ensuite, il retire toutes les lettres ‘X’ :
5’-GGATGXXXXXXXXXGTCA-3’
3’-CCTACXXXXXXXXXXXXX-5’
Des doigts de zinc
On a découvert en 1987, un motif protéique naturel baptisé doigt de zinc. On y trouve une hélice-alpha accouplé à un feuillet-bêta antiparallèle via un ou plusieurs atomes de zinc. Quatre acides aminés (2 histidines et 2 cystéines) prennent en sandwich ces atomes de zinc selon une géométrie tétraédrique. Chaque doigt de zinc permet de reconnaître des séquences de nucléotides (par série de trois). En combinant 6 à 8 doigts de zinc, on obtient des protéines spécifiques. Il suffit pour cela d’une vingtaine de paires de bases. En 1996, on a eu l’idée géniale de coupler de manière artificielle un doigt de zinc à la protéine FokI. L’idée était d’obtenir des nucléases à doigt de zinc (acronyme ZFN pour « Zinc Finger Nucléase »).
On peut ainsi cibler des séquences génétiques de manière spécifique. Une fois ciblées, le clivage s’opère grâce à l’action de la nucléase associée. La technique a d’abord été testée pour créer de nouvelles variétés de soja plus résistantes à la sécheresse. Puis, on induit la tolérance à un herbicide pour le tabac ou le maïs. Les pétunias sont, eux, génétiquement modifiés pour des raisons esthétiques. En 2002, on teste la technique ZFN sur un animal vivant. Le problème des ZFNs est qu’elles nécessitent plusieurs mois d’expérimentation. Ceci est un handicap certain dans la course frénétique aux brevets que se livrent les industriels impliqués dans l’ingénierie génomique.
Des activateurs de transcription
En 2009, on découvre les TALE (acronyme pour transcription activator-like effector). Il s’agit d’effecteurs de type activateur de transcription produits par les bactéries pathogènes du genre Xanthomonas. On y trouve des répétitions d’un motif de 34 acides aminés. Au sein de ces répétitions, les acides aminés n°12 et n°13 reconnaissent un nucléotide spécifique selon le code suivant :
NI = A ; HD = C ; NK/NH = G et NG = T (où N est l’asparagine ; I l’isoleucine ; H l’histidine ; D l’aspartate et K la lysine)
Les TALENs (acronyme pour nucléases effectrices de type activateur de transcription) sont ainsi apparus en 2010. Elles ont permis de ramener le temps nécessaire aux expérimentations à seulement quelques semaines. Comme les ZFNs, Les TALENs sont des protéines chimériques de type ciseaux moléculaires. On y trouve un domaine de liaison à l’ADN où le TALE a remplacé le doigt de zinc. Ce domaine est toujours associé à une endonucléase de type FokI. Une fois introduites dans les cellules, ces TALENs offrent la possibilité d’éditer, sur place, les génomes de nombreux organismes modèles. Ceci, de manière rapide, spécifique et efficace.
Depuis leur première utilisation, on a largement employé les TALENs, aussi bien in vitro qu’in vivo. Les cibles étaient des levures, des plantes, des poissons. Furent aussi modifiés, le xénope, la drosophile, le ver Caenorhabditis elegans. Puis on modifia le rat, la souris, le lapin, la vache ou le cochon. Toutefois, avoir des temps d’expérimentation qui se mesurent en semaines est encore trop lent pour des industries aussi compétitives.
Des ciseaux moléculaires
C’est dans ce contexte que l’on découvre en 2011 une protéine du système immunitaire adaptatif de la bactérie Streptococcus pyogenes. Cette protéine permet de reconnaître et d’éliminer l’ADN étranger des virus bactériophages. La clé est la présence de répétitions en grappes palindromiques courtes régulièrement espacées de séquences virales appelées CRISPR. Cet acronyme signifie Clustered Regularly Interspaced Short Palindromic Repeats. Associée à ces répétitions, on trouve aussi une méganucléase. Il s’agit d’une endonucléase spécialisée dans la coupure de l’ADN et qui est guidée par un ARN. Il y a deux zones de coupe actives. Une pour chaque brin de la double hélice, situées l’une en face de l’autre.
La protéine complète s’appelle Cas9 (acronyme pour CRISPR associated protein 9). Elle sera, dès 2012, brevetée pour devenir un outil de bidouillage du génome. Cet outil est en effet capable d’altérer n’importe quel morceau d’ADN ou d’ARN de manière parfaitement ciblée. Par rapport aux ZFNs ou aux TALENs, Cas9 a gagné en popularité. Ceci en raison de sa capacité à couper l’ADN précisément à n’importe quel emplacement complémentaire de son ARN guide. Le ciblage de l’ADN par Cas9 est ainsi direct. Surtout il ne nécessite pas de modification de la protéine, mais seulement une modification de l’ARN guide.
Pour utiliser ces nouveaux ciseaux génétiques CRISPR/Cas9 on doit donc fabriquer un ARN guide. Celui-ci va venir se fixer sur les brins d’ADN de la cible de manière complémentaire au moyen d’une tête chercheuse. Une fois la tête chercheuse fixée, l’ARN utilise sa queue afin d’attirer à lui la protéine-ciseau Cas9. Cette dernière creuse ainsi un trou dans la séquence d’ADN ciblée.
Réparations
Afin de réparer cette cassure, la cellule peut faire appel à un système de réparation peu fiable appelé NHEJ. Cet acronyme signifie non-homologous end joining ou réparation par jonction d’extrémités non homologues. La jonction d’extrémités non homologues lie les extrémités cassées entre elles. Elle peut s’accompagner de la perte ou du gain de quelques nucléotides. Ceci empêche bien sûr le gène de s’exprimer.
C’est de cette manière que les chercheurs invalident l’expression d’un gène afin de mieux comprendre son fonctionnement. Ce système d’invalidation d’un gène est extrêmement efficace. Il marche quasiment à 100 %. Il est aussi très rapide, et peu cher. La cellule peut aussi utiliser la réparation par recombinaison homologue. Elle utilise pour cela une séquence d’ADN homologue qui sert de modèle pour la restauration de l’intégrité de l’ADN génomique. Ainsi, la réparation peut être précise et contrôlée. La copie insérée est identique à la séquence de l’ADN qui sert de source pour la recombinaison. Ce deuxième système de modification avec réparation est plus délicat. Toutefois, on sait l’utiliser depuis 2015 pour modifier le génome d’embryons humains ou pour lutter contre le cancer.
On peut donc se poser la question si le SARS-CoV-2 ne serait pas issu d’un laboratoire trafiquant les génomes cellulaires. Car ce ne sont pas les outils qui manquent : ZFNs, TALENs ou CRISPR/Cas9. La production scientifique dans ce domaine a en effet quadruplé entre 2011 et 2015 dans le monde. En Chine, elle a été multipliée par 15. La technique a donc incontestablement pris une position dominante dans ce domaine scientifique et ses applications. Le 17 mars 2016 l’Institut Pasteur de Shanghai (IPS) et l’Institut de virologie de Wuhan (WIV) ont uni leurs efforts. Ceci dans un but de recherche afin de mieux répondre aux grands enjeux de santé publique.
Virus chimériques
La finalisation des travaux du laboratoire de haute sécurité biologique « P4 » de Wuhan était claire. On investissait dans la lutte contre les virus hautement pathogènes et à haute dangerosité. Le feu a été mis aux poudres, par 2 chercheurs de ce fameux laboratoire WIV, Zhengli-Li Shi et Xing-Yi Ge. Dans un article de décembre 2015, on peut en effet lire :
« En utilisant le système génétique inverse du SARS-CoV, nous avons généré et caractérisé un virus chimère exprimant la protéine-S du virus SHC014 de la chauve-souris dans un génome SARS-CoV adapté à la souris. Les résultats obtenus montrent que les virus du groupe 2b SHC014 encodant la nouvelle protéine-S dans un génome sauvage peuvent utiliser de manière efficace de multiples orthologues du récepteur ACE2 humain impliqué dans le SRAS et se reproduire dans les cellules du système respiratoire primaire humain afin de produire in vitro des titres équivalents à ceux rencontrés dans les épidémies de SARS-CoV. De plus, des expériences in vivo démontrent la réplication du virus chimère dans les poumons de la souris avec des effets pathogènes évidents.
Une évaluation des modalités prophylactiques et des thérapies immunitaires ciblant le SRAS actuellement disponible a révélé une très faible efficacité puisque les approches vaccinales ou par anticorps monoclonaux n’ont pas réussi à neutraliser ni à protéger de l’infection par les CoVs dotés de la nouvelle protéine-S. Sur la base de ces résultats, nous avons réussi à refabriquer de manière synthétique un virus recombinant complet de type SHC014 qui a démontré des capacités robustes de réplication à la fois in vitro et in vivo. Ceci suggère un risque de ré-émergence du SARS-CoV à partir des virus actuellement en circulation dans les communautés de chauve-souris. »
Nature Medicine (Vol. 21, n°12, pp.1508-1514)
Naturel ou synthétique ?
On ne saurait être plus clair sur les capacités de ces laboratoires P4 à faire joujou avec des virus chimériques. Ils sont libres de créer des virus de type SARS-CoV totalement artificiels et potentiellement très pathogènes. On notera aussi une kyrielle de co-auteurs américains. Ceci démontre que ces apprentis sorciers sont plus américains (13 chercheurs) que chinois (2 chercheurs).
Toutes les analyses génétiques du SARS-CoV-2 montrent des similitudes très élevées avec les coronavirus qui infectent les chauves-souris. Toutefois, les protéines-S de ces virus « naturels » ne peuvent se fixer au récepteur humain ACE2. En revanche, des protéines-S de coronavirus circulant dans le génome des pangolins peuvent se fixer sur les récepteurs ACE2 humains. Grâce aux ciseaux moléculaires, on pourrait donc, en théorie, recréer en laboratoire un virus chimérique. Celui-ci marierait le génome des coronavirus actifs chez la chauve-souris avec la protéine-S des virus actifs chez le pangolin. On obtiendrait ainsi un génome très similaire à celui observé dans l’épidémie actuelle.
La question est donc bien ouverte. Elle mérite sûrement mieux qu’un simple haussement d’épaules. Elle gêne la langue de bois des ambassadeurs chinois. Cette question jette enfin un doute sérieux sur les insinuations américaines. Nous essaierons dans la prochaine chronique d’y voir plus clair.
Par Marc HENRY


Leave a Reply